
王斌课题组在Environ Sci Technol期刊发表观点论文探讨小样本学习用于下一代人群健康风险评估的方法、挑战和展望
导读:
在环境暴露与人群健康研究中,传统流行病学研究面临着诸多挑战,如高维数据难以处理、大样本需求与研究成本之间的矛盾等。这些限制在应对复杂暴露环境时尤为显著。本研究提出,小样本学习作为一种创新方法,可以通过整合暴露组数据、人工智能技术和系统生物学信息,显著提高评估效率和结果的可解释性。文章详细讨论了大规模暴露组数据库的应用、多组学数据整合的必要性,以及深度学习技术在优化小样本学习中的潜力与实践。未来,基于AI和大数据的风险评估方法有望成为解决传统流行病学局限的关键路径。这项研究由北京大学王斌教授课题组与何松教授(军事医学科学院)、张乐教授(电子科技大学)和方明亮教授(复旦大学)多方合作完成。
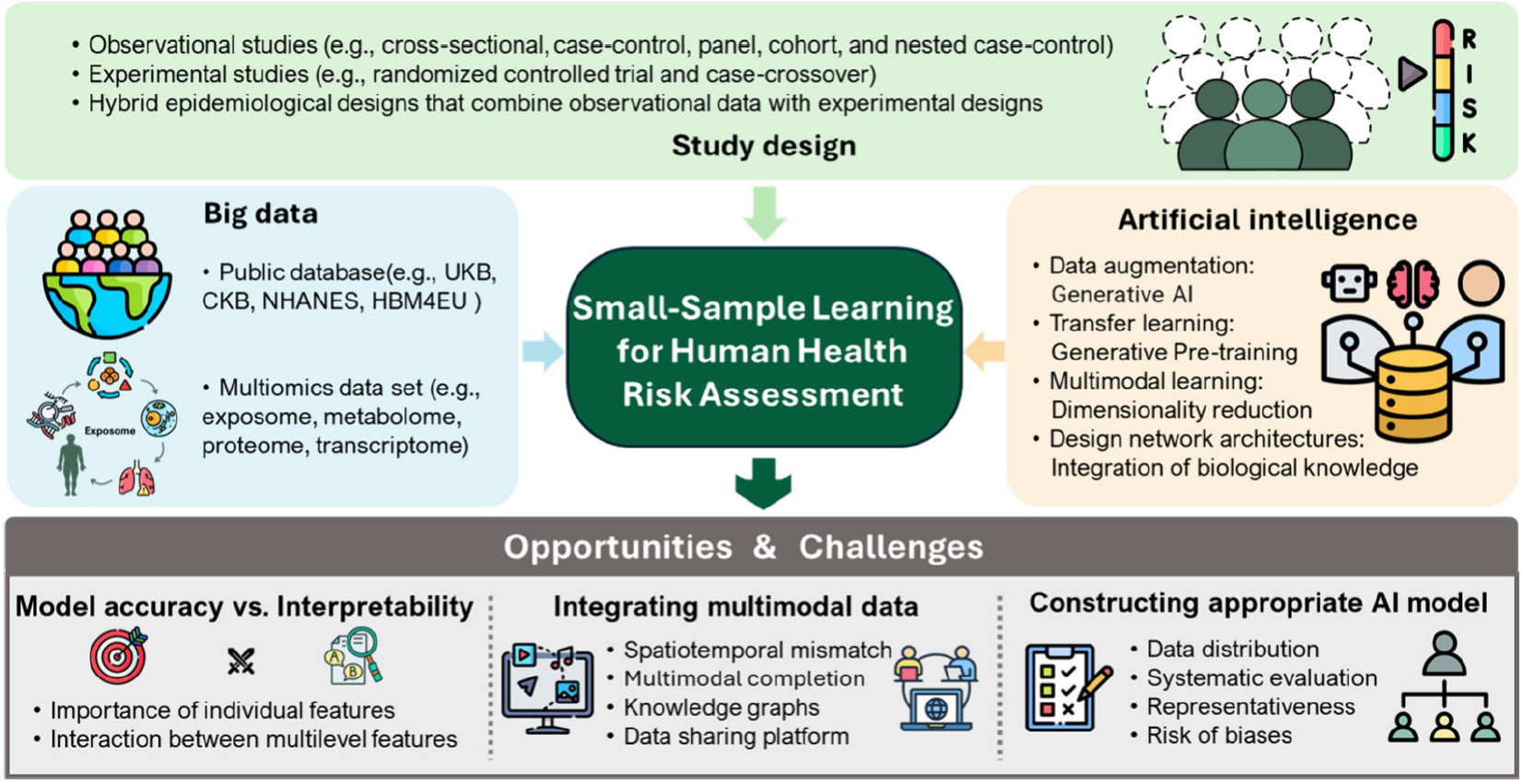
图1. 小样本机器学习用于人群健康风险评价总结
英文题目:Small-Sample Learning for Next-Generation Human Health Risk Assessment: Harnessing AI, Exposome Data and Systems Biology
原文链接:https://doi.org/10.1021/acs.est.4c11832
第一单位:北京大学
期刊名称:Environmental Science & Technology (中科院1区)

涉及环境暴露的人类健康风险评估(HRA)面临许多挑战,尤其是在运用传统流行病学方法处理大规模暴露组数据时,往往存在高维数据和分析精度的难题。这些数据包括了外源性环境因素(如污染物、金属、噪声等)和内源性生物信息(如基因组、转录组、肠道微生物群等)1。例如,人体暴露组数据库(HExpMetDB)包含了超过2万种化学物质,并根据风险筛选出了7,770种化学物质2。我们最近开发的暴露组数据库(ExposomeX,http://www.exposomex.cn/#/database101)整合了约1.19亿种暴露数据及17,186种疾病亚型。此外,我们还构建了TOXRIC数据库(https://toxric.bioinforai.tech/),该数据库全面整合了毒理学数据,涵盖113,372种化学物质,涉及13个毒性类别、1,474个毒性终点(包括体内和体外终点),以及39种分子特征,提供标准化属性数据、实用基准测试、分子表征可视化及直观功能界面3。然而,揭示因果关系的过程需要大量时间和资源,包括大样本量、长期随访和高成本的实验分析。为了解决这一问题,小样本学习成为一种潜在的有效方法,尤其适用于样本招募困难或分析成本高昂的情况。通过从小样本数据中提取有意义的信息,可以显著提高HRA效率,特别是在评估新污染物或个体易感性差异的场景中。随着大数据和人工智能技术的快速发展,如何基于高维数据进行可靠的统计分析,成为一个亟待解决的难题。未来,结合大数据和AI技术的HRA模型将能更加精准地筛查风险因素、量化效应值,并为复杂环境暴露场景中的疾病负担提供量化数据。本观点文章强调,优化环境健康风险评估和改善公共健康的关键在于选择恰当的流行病学研究设计,同时结合大数据和AI技术开发创新策略(详见图1)。
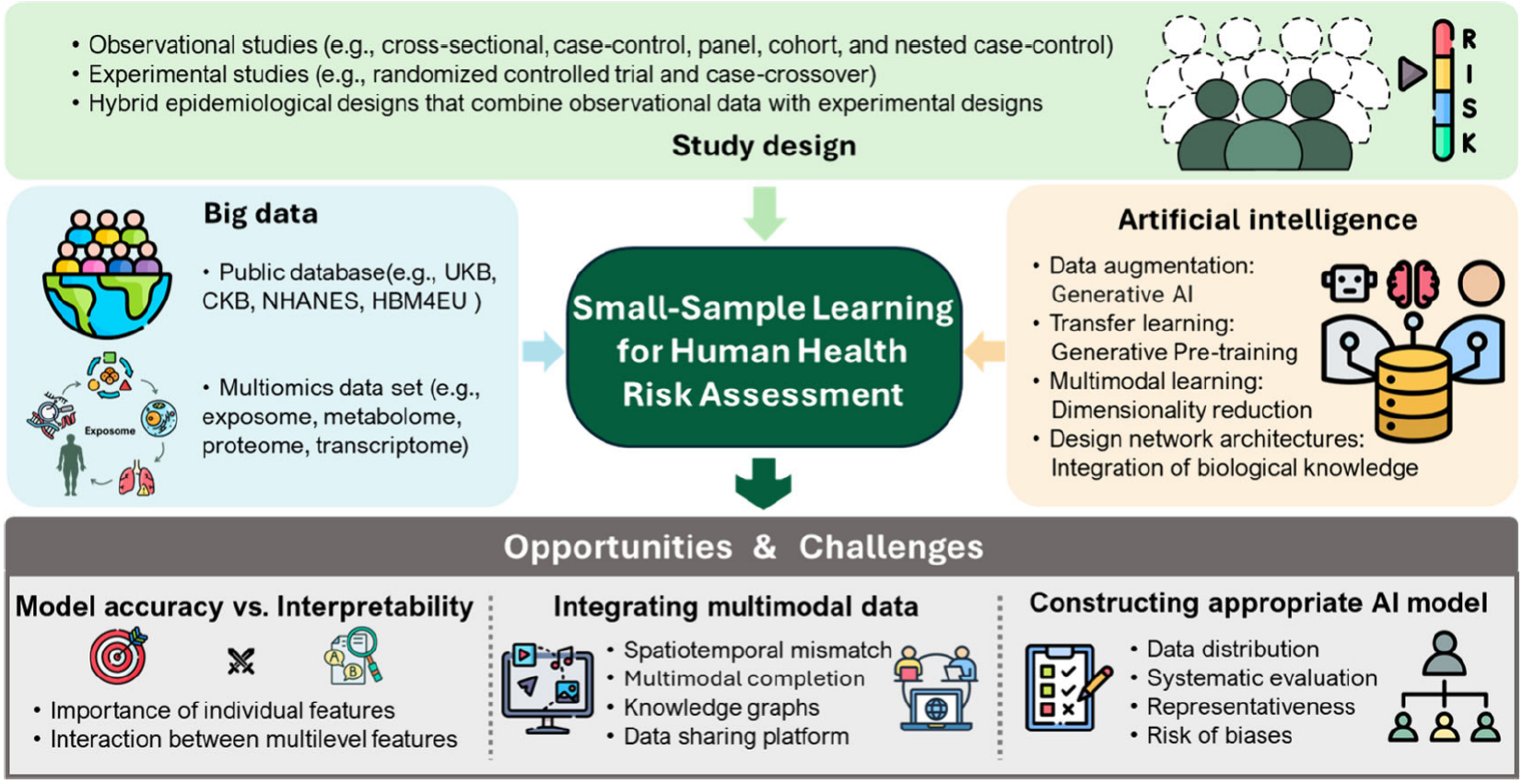
图1. 环境暴露健康风险评估(HRA)中的小样本学习应用。HRA建模需要考虑研究设计、大数据整合和AI模型三个方面。未来小样本学习的发展应该优先考虑平衡模型的准确性和可解释性,有效地整合多模态数据,并构建泛化能力强的AI模型,在解决数据偏倚的同时结合先验知识。
针对以上研究需求,以下三个方法可促进此类研究的开展,并归纳未来的机遇和挑战,具体如下:
■研究方法
1、选择合适的流行病学设计对于平衡证据强度、样本量和可用资源至关重要。
在环境暴露相关的HRA中,可以选择不同的流行病学研究设计来满足不同的研究目标和数据特性。这些设计包括观察性研究(如横断面研究、病例对照研究、队列研究)和实验性研究(如随机对照试验、病例交叉研究)。此外,还可以结合观察性数据与实验/准实验性设计,采用混合流行病学设计,以增强研究证据的强度,例如倾向评分匹配和工具变量法。如果研究目标是评估某些健康结局的发生率,前瞻性设计(如队列研究或纵向研究)通常更为适用;而在难以招募更多参与者的情况下,采用重复测量的方法可以有效提高数据利用率。此外,针对关键时间窗口和敏感人群的已有信息,有助于合理规划样本量,从而开发更加精确的HRA模型。例如,Stelzer等人对63名自然分娩女性进行了纵向研究,采集了代谢组、蛋白质组和免疫组三种组学数据,结合堆叠泛化(SG)模型,成功实现了对分娩时间的精确预测4。
2、充分利用暴露组大数据。主要体现在以下两个关键方面:
(1)利用大规模且可靠的人类生物监测数据库,提供环境暴露与健康结局关联的先验知识。过去几十年,许多国家和地区启动了大规模的人类生物监测项目,旨在探究生活方式、环境因素和遗传因素如何影响人类疾病。这些项目推动了数据的开放和资源共享,提供了公平透明的访问机制,为研究人员提供了宝贵的资源。比如,UK Biobank (UKB)、欧洲人类生物监测计划 (HBM4EU)、美国国家健康与营养调查 (NHANES) 和中国健康与养老追踪调查 (CHARLS) 等数据库,都涵盖了生物样本、遗传学、成像、健康记录、生活方式、空气污染和化学暴露等多方面的信息。这些资源为验证环境暴露对疾病影响的假设提供了重要支持。例如,Argentieri 等人利用 UK Biobank (UKB) 的蛋白质组学数据(45,441人),从2,897种蛋白质中筛选出204种关键血浆蛋白,成功构建了一个预测生物年龄的蛋白质组时钟5。该模型在中国(3,977人)和芬兰(1,990人)等独立队列中进行了验证,显示出较高的预测准确性。通过在大人群中识别出具有强泛化性的生物标志物,该方法能够在小规模队列中实现精准的风险评估,不仅能提高对敏感人群的识别能力,还能减少误差,提升结果的可解释性。在一些人类生物监测数据较为稀缺的情况下,体外-体内外推(IVIVE)方法也是一种有效的工具。最近,Han 等人总结并探讨了下一代HRA中的 IVIVE 方法,其中包括扩展IVIVE的适用范围,例如考虑母体化合物及其代谢物的联合风险;以及将系统生物学、多组学技术和不良结局网络(AOP)等新兴工具整合到IVIVE中,以实现更深入、更全面的风险评估6。
(2)将多组学数据引入小样本学习。环境暴露通常通过多种途径影响生物过程,最终可能导致不良健康结果。但多组学研究面临一些挑战,特别是高成本和数据的高维度问题。以单细胞转录组学为例,单个人的测量费用可能高达1,500到2,000美元,这使得在高通量组学评估中,样本量通常较小。此外,随着数据维度的增加,模型需要处理的特征组合也会急剧增加,这种“维度灾难”使得某些特征组合变得非常稀疏,甚至完全缺失,影响了模型的学习和泛化能力。不同的组学数据还可能使用不同的测量技术,这会导致它们在特征和分布上有所不同。在这种情况下,深度学习算法能够帮助我们解决一些问题,它能自动捕捉数据中的复杂非线性关系,并从质量较低的数据中提取出高质量的特征表示,还可以进行降维以减少高维度带来的挑战7。例如,Cao等人提出的GLUE框架使用知识图谱将不同组学数据的特征空间进行整合8。在该框架中,节点代表不同组学的特征,边则表示它们之间的调控关系。这个模型能够很好地处理单细胞多组学数据,并且在样本较少的情况下依然表现得很稳定。为了减轻多组学数据中的多重共线性问题,我们需要确保数据质量高,且包含足够多样的特征。同时,选择合适的模型和正则化策略,并结合现有的生物学知识(如基因调控网络和蛋白质相互作用)来进一步提升模型的效果。
3、充分利用深度学习方法在解决小样本问题上的巨大潜力。
尽管大数据时代为我们提供了丰富的资源,如大规模生物监测数据库、可穿戴设备、生物成像和多组学数据,但在环境健康风险评估(HRA)研究中,整合来自不同来源的信息仍然面临很多挑战。对于小样本学习,我们可以采用以下四种常见方法:
(1)数据增强。数据增强是一种通过生成与真实数据高度相似的新数据来扩展数据集的技术。在有限的标注数据下,AI生成内容(AIGC)能够利用大量未标注数据学习数据分布,生成图像、文本、音频和视频等样本,从而提高模型的泛化能力。常见的生成模型包括:生成对抗网络(GANs):通过生成器和判别器的对抗训练,GANs能够生成逼真的样本,利用潜在空间的灵活性有效捕捉数据的特征;扩散模型:通过逐步去噪的过程从随机噪声中生成高质量的数据,尤其适合在小样本条件下输出多样化的样本;变分自编码器(VAEs):VAEs通过编码器将数据压缩至潜在空间,再通过解码器生成新样本。它提供了一个稳定的生成过程,能够保持数据的多样性和结构完整性。AIGC技术已经在分子属性预测9和药物设计10等领域取得了显著进展,但在HRA相关数据集中的应用尚未得到充分探索。这为将AIGC方法应用于环境健康风险评估(HRA)提供了一个新的研究方向。
(2)迁移学习。通过在大型通用数据集上训练机器学习或深度学习模型,然后在小型特定领域数据集上进行微调,迁移学习已经成为HRA研究中的重要方法之一。它的关键应用包括利用化学物质的结构活性关系和疾病间的共享生物网络,将研究从常见污染物扩展到新兴污染物,从常见疾病迁移到罕见疾病。在应用迁移学习时,需要对源和目标群体的关键因素进行对齐,比如暴露特征、人口结构、疾病发生率和类型。这种对齐可以提高模型的适用性和准确性。微调是迁移学习中的核心环节,包括调整分层学习率、冻结模型的早期层并只微调后期层,以及使用正则化技术防止过拟合。生成式预训练(GPT)模型通过注意力机制和无监督预训练在自然语言处理领域取得了开创性成果。受此启发,研究人员将自监督预训练技术应用到大规模单细胞转录组数据上,开发出适用于小型患者数据集的基础模型,用于识别疾病治疗靶点11。在未来,类似ChatGPT的模型可能会被训练用于分析人类生物监测数据库,但这些应用还需要进一步验证和优化。
(3)多模态学习。在HRA研究中,暴露数据主要包括数值向量(如多组学数据、污染物浓度)、图形(如分子图、生物知识图谱)、文本(如电子健康记录、蛋白序列)、视觉数据(如成像、遥感)和音频数据(如环境噪声)。在系统生物学任务中,多模态学习通过整合不同模态的互补信息,不仅能提升小样本学习的性能,还能为环境健康问题提供更全面的视角12。多模态学习可以通过多种架构实现,其中一种常见的方法是对不同模态数据进行联合建模。这可以通过设计专门的架构,或开发将模态间相似概念映射到共享潜在空间的基础模型,从而生成统一的内部表征。例如,将遥感图像、传感器检测的污染物浓度数据和事件文本描述整合起来,以输出特定任务的结果。这类基础模型已成功应用于单细胞组学数据8和生物医学数据整合领域13。另一种方法是构建跨模态的知识图谱(KG)。在系统生物学中,多层特征可以表示为异构图,通过捕捉化学物质、基因、蛋白质和疾病之间的关联,利用图神经网络(GNN)挖掘这些实体间的潜在关系。多模态模型为整合外暴露与内暴露生物标志物提供了一种新思路,可以生成统一的特征向量,全面反映个体的暴露特征。
(4)特殊的网络架构或算法。基于生物信息设计的稀疏网络结构可以有效减少模型参数。例如,DCell模型利用神经网络中的隐藏节点模拟特定的细胞过程和功能14,而P-NET模型则通过基因与通路的关联来构建网络15。这些简化的网络架构不仅提升了小样本学习的精度,还能量化网络中生物过程的重要性,从而增强模型的可解释性。另一种方法是深度森林模型(DF),它是一种多层树状结构的级联模型,超参数较少,能够自适应调整模型复杂度。深度森林在小规模数据集中表现尤其出色,非常适合小样本学习任务16。此外,受AOP框架的启发,可以将环境暴露、分子起始事件(MIE)、关键分子事件(KE)和不良结局整合到网络架构中。通过逐步增加网络的复杂性,不仅能够捕捉关键生物学过程,还能更系统地分析环境暴露对健康的影响17。
需要注意的是,在特定的小样本环境研究中,这些方法通常不会单独使用,而是相互结合形成系统性解决方案。例如,Huang等人整合了基因组数据、细胞信号、基因表达水平和临床记录,构建了生物医学知识图谱,并进行自监督预训练18。通过结合多模态学习和迁移学习,他们的模型在严格的零样本条件下,成功预测了多种疾病的药物适应症和禁忌症,包括一些目前尚无治疗药物的疾病。
■机遇与挑战
1、平衡模型的准确性与可解释性。
尽管深度学习在许多领域取得了巨大成功,但其可解释性依然是一个难题。在HRA研究中,将模型的权重和参数转化为有意义的生物学信息尤为重要。这不仅能为医疗服务提供可靠依据,还能为疾病的发生和发展提供潜在的生物学解释。常见的可解释性方法包括SHAP和Transformer架构中的注意力权重。然而,在小样本数据场景中,依赖数据驱动的解释有时可能导致虚假的关键特征,偏离实际的生物学规律19。另一种方法是通过对特征进行计算扰动(如删除、随机遮掩或组学数据反转),评估与疾病相关的关键环境暴露和生物学特征,并量化这些扰动对预测结果的影响。相比随机特征,扰动已知特征(如MIE和KE)通常更能增强解释性,但这需要依赖毒性通路的先验知识。上述方法主要用于评估单个特征的重要性,而深度学习的优势在于捕捉特征之间的相互作用。例如,可以将环境暴露、生物过程和疾病进程划分到模型的不同层次,并通过分析各层神经元的激活,识别环境暴露与生物效应之间的非线性交互。扰动特征嵌入还可用于评估潜在的交互作用,例如扰动环境暴露特征的嵌入,观察其对生物效应嵌入的影响,从而识别环境暴露的靶标。这种方法已被用于发现基因间的相互作用,但通常需要较大的计算资源11。此外,通过同时扰动两个或多个环境暴露特征,可以评估它们对疾病或生物效应的联合影响,进而分析环境暴露的混合效应。为了提高模型的可解释性,还可以结合主动学习和精心设计的查询策略,通过优先验证不确定性较高或影响较大的未标注样本,不仅降低了标注成本,还提升了模型的解释能力。
2、 整合多模态数据
目前,AI在HRA中的应用主要利用单一数据类型解决单一任务。然而,未来的发展方向应该是整合多模态数据,全面研究环境暴露如何通过不同层次的生物过程诱发疾病。随着多模态数据量的增加,迫切需要开发适合的框架来整合结构化和非结构化数据。这些框架既要生成准确的特征表示,又要保留每种数据类型中的生物学信息。整合外源组数据面临的一个难点在于,不同类型的数据往往有不同的分辨率和时间尺度。例如,生物样本中的污染物测定、基因表达或代谢物数据通常只反映一个特定时间段的暴露情况,而健康结果(如慢性疾病)可能需要更长时间才会显现,二者在时间上并不匹配。此外,多模态数据还存在样本缺失的问题。由于不同类型数据是互补的,已知的数据可以用来预测和填补缺失的部分,从而提高数据的完整性。基于知识图谱(KG)的方法也为多模态数据的整合提供了新的思路。KG能够为外源组数据的融合提供更深入的生物学见解,但目前这一方法还处于初步阶段,图谱构建和推理算法需要进一步优化。因此,建设高质量的暴露组数据共享平台显得尤为重要。目前虽然已有一些大型的高质量数据库,但大量数据仍分散在文献中,数据来源、格式和样本量不统一。将这些数据整合和标准化,是创建高质量开放数据平台(如ExposomeX和TOXRIC)的关键步骤,从而推动数据共享和利用的进一步发展。
3、 构建具有更强泛化能力的AI模型
在选择算法之前,必须采取多种策略应对小样本数据带来的挑战。即使是性能最优的模型,在数据质量欠佳的情况下也难以获得有意义的知识。解决方法包括:主动收集和整理多样化的数据来源(例如,穿戴设备数据和连续的环境监测数据),并推动数据标准化和处理流程自动化;全面分析小样本的分布,以减少偏倚;结合生物学和环境化学知识进行特征工程;清理数据中的噪声和异常值,同时妥善处理缺失数据。在选择模型算法时,除了追求准确性外,还需权衡模型的复杂性、鲁棒性、泛化能力和可解释性。通过系统评估,可以确保这些因素之间的平衡。在利用大规模人类生物监测数据库进行迁移学习时,应特别注意可能因人群选择产生的潜在偏倚。此外,将现有的生物学知识融入模型设计,是解决小样本问题的一种有前景的途径。在模型开发完成后,需进一步评估小样本数据在HRA中的代表性,并仔细考虑潜在偏倚带来的风险,以确保研究结果的可靠性和实用性。
参考文献:
1. Fang, M. L.; Hu, L. G.; Chen, D.; Guo, Y. M.; Liu, J. M.; Lan, C. X.; Gong, J. C.; Wang, B., Exposome in human health: Utopia or wonderland? Innovation-Amsterdam 2021, 2, (4), 100172.
2. Zhao, F. R.; Li, L.; Chen, Y.; Huang, Y. C.; Keerthisinghe, T. P.; Chow, A.; Dong, T.; Jia, S. L.; Xing, S. P.; Warth, B.; Huan, T.; Fang, M. L., Risk-Based Chemical Ranking and Generating a Prioritized Human Exposome Database. Environ Health Persp 2021, 129, (4), 47014.
3. Wu, L.; Yan, B.; Han, J.; Li, R.; Xiao, J.; He, S.; Bo, X., TOXRIC: a comprehensive database of toxicological data and benchmarks. Nucleic acids research 2023, 51, (D1), D1432-d1445.
4. Stelzer, I. A.; Ghaemi, M. S.; Han, X.; Ando, K.; Hédou, J. J.; Feyaerts, D.; Peterson, L. S.; Rumer, K. K.; Tsai, E. S.; Ganio, E. A.; Gaudillière, D. K.; Tsai, A. S.; Choisy, B.; Gaigne, L. P.; Verdonk, F.; Jacobsen, D.; Gavasso, S.; Traber, G. M.; Ellenberger, M.; Stanley, N.; Becker, M.; Culos, A.; Fallahzadeh, R.; Wong, R. J.; Darmstadt, G. L.; Druzin, M. L.; Winn, V. D.; Gibbs, R. S.; Ling, X. B.; Sylvester, K.; Carvalho, B.; Snyder, M. P.; Shaw, G. M.; Stevenson, D. K.; Contrepois, K.; Angst, M. S.; Aghaeepour, N.; Gaudillière, B., Integrated trajectories of the maternal metabolome, proteome, and immunome predict labor onset. Science Translational Medicine 2021, 13, (592), eabd9898.
5. Argentieri, M. A.; Xiao, S.; Bennett, D.; Winchester, L.; Nevado-Holgado, A. J.; Ghose, U.; Albukhari, A.; Yao, P.; Mazidi, M.; Lv, J.; Millwood, I.; Fry, H.; Rodosthenous, R. S.; Partanen, J.; Zheng, Z.; Kurki, M.; Daly, M. J.; Palotie, A.; Adams, C. J.; Li, L.; Clarke, R.; Amin, N.; Chen, Z.; van Duijn, C. M., Proteomic aging clock predicts mortality and risk of common age-related diseases in diverse populations. Nature Medicine 2024, 30, (9), 2450-2460.
6. Han, P.; Li, X.; Yang, J.; Zhang, Y.; Chen, J., Advancing Toxicity Predictions: A Review on in Vitro to in Vivo Extrapolation in Next-Generation Risk Assessment. Environment & Health 2024, 2, (7), 499-513.
7. Hu, Y.; Wan, S.; Luo, Y.; Li, Y.; Wu, T.; Deng, W.; Jiang, C.; Jiang, S.; Zhang, Y.; Liu, N.; Yang, Z.; Chen, F.; Li, B.; Qu, K., Benchmarking algorithms for single-cell multi-omics prediction and integration. Nature Methods 2024, 21, 2182-2194.
8. Cao, Z.-J.; Gao, G., Multi-omics single-cell data integration and regulatory inference with graph-linked embedding. Nature Biotechnology 2022, 40, (10), 1458-1466.
9. Wang, Y.; Wang, J.; Cao, Z.; Barati Farimani, A., Molecular contrastive learning of representations via graph neural networks. Nature Machine Intelligence 2022, 4, (3), 279-287.
10. Igashov, I.; Stärk, H.; Vignac, C.; Schneuing, A.; Satorras, V. G.; Frossard, P.; Welling, M.; Bronstein, M.; Correia, B., Equivariant 3D-conditional diffusion model for molecular linker design. Nature Machine Intelligence 2024, 6, (4), 417-427.
11. Theodoris, C. V.; Xiao, L.; Chopra, A.; Chaffin, M. D.; Al Sayed, Z. R.; Hill, M. C.; Mantineo, H.; Brydon, E. M.; Zeng, Z.; Liu, X. S.; Ellinor, P. T., Transfer learning enables predictions in network biology. Nature 2023, 618, (7965), 616-624.
12. Liu, W.; Chen, J.; Wang, H.; Fu, Z.; Peijnenburg, W. J. G. M.; Hong, H., Perspectives on Advancing Multimodal Learning in Environmental Science and Engineering Studies. Environmental Science & Technology 2024, 58, (38), 16690-16703.
13. Moor, M.; Banerjee, O.; Abad, Z. S. H.; Krumholz, H. M.; Leskovec, J.; Topol, E. J.; Rajpurkar, P., Foundation models for generalist medical artificial intelligence. Nature 2023, 616, (7956), 259-265.
14. Ma, J.; Yu, M. K.; Fong, S.; Ono, K.; Sage, E.; Demchak, B.; Sharan, R.; Ideker, T., Using deep learning to model the hierarchical structure and function of a cell. Nature Methods 2018, 15, (4), 290-298.
15. Elmarakeby, H. A.; Hwang, J.; Arafeh, R.; Crowdis, J.; Gang, S.; Liu, D.; AlDubayan, S. H.; Salari, K.; Kregel, S.; Richter, C.; Arnoff, T. E.; Park, J.; Hahn, W. C.; Van Allen, E. M., Biologically informed deep neural network for prostate cancer discovery. Nature 2021, 598, (7880), 348-352.
16. Wu, L.; Gao, J.; Zhang, Y.; Sui, B.; Wen, Y.; Wu, Q.; Liu, K.; He, S.; Bo, X., A hybrid deep forest-based method for predicting synergistic drug combinations. Cell Reports Methods 2023, 3, (2), 100411.
17. Ciallella, H. L.; Russo, D. P.; Aleksunes, L. M.; Grimm, F. A.; Zhu, H., Revealing Adverse Outcome Pathways from Public High-Throughput Screening Data to Evaluate New Toxicants by a Knowledge-Based Deep Neural Network Approach. Environmental Science & Technology 2021, 55, (15), 10875-10887.
18. Huang, K.; Chandak, P.; Wang, Q.; Havaldar, S.; Vaid, A.; Leskovec, J.; Nadkarni, G. N.; Glicksberg, B. S.; Gehlenborg, N.; Zitnik, M., A foundation model for clinician-centered drug repurposing. Nature Medicine 2024, 30 (12), 3601-361.
19. Li, H.; Yin, N.; Yang, R.; Faiola, F., Advancing Environmental Toxicology In Vitro: From Immortalized Cancer Cell Lines to 3D Models Derived from Stem Cells. Environment & Health 2024, 2, (6), 332-349.
作者介绍:
伍天翔(第一作者)

北京大学公共卫生学院博士研究生。主要研究方向:人工智能和计算毒理学。以第一作者在环境领域国际权威期刊Environmental Science & Technology发表论文2篇,在中文核心期刊《科学通报》上发表论文1篇。
王斌(通讯作者)

研究员/长聘副教授,北京大学生育健康研究所/北京大学城市与环境学院(兼聘)。研究方向:环境健康、暴露组学、人工智能。主持国家自然科学面上和青年基金4项,骨干参与国家重点研发项目3项。以第一或通讯作者在Science、Environ Health Persp、Environ Sci Tech、The Innovation等国际权威期刊发表长篇及评述论文共60篇(H指数=45,他引6000余次)。担任环境健康领域TOP期刊Environ Sci Tech副主编。开设本科生和研究生教改课程“暴露组学”,全球健康国际高端公共卫生硕士课程“Environment & Health”。担任中国队列共享平台“环境与人群健康”组长,环境诱变剂学会环境与生育健康专委会副秘书长。获得北京预防医学会科技二等奖、华夏医学科技奖三等奖和“全国科技系统抗击新冠疫情优秀个人”称号。

