
ES&T | 开发快速构建“暴露–生物学–疾病”关联的暴露组学分析平台
暴露组学已成为下一代环境健康研究的核心方向之一,但目前尚缺乏可对暴露组数据进行标准化分析的有效平台。本研究提出了一个暴露组分析框架,并开发了一个新型整合平台 ExposomeX,以加速“暴露–生物学–疾病”关联的发现。该平台包含六大核心功能模块:暴露组数据库(E-DB)、生物关联分析(E-BIO)、统计分析(E-STAT)、质谱数据处理(E-MS)、荟萃分析(E-META)、数据可视化(E-VIZ)。其中,E-DB主要通过整合已有数据库和高质量、同行评审的高质量文献和数据库构建,涵盖暴露物、疾病及其相关分子和通路信息;E-BIO基于这些数据建立暴露与疾病之间的生物学关联;E-META利用同一数据源对特定暴露与疾病之间的关系进行定量评估;E-STAT集成了稳健可靠的模型,用于统计分析;E-MS支持非靶向分析数据的统计处理和注释;E-VIZ则用于高维数据的可视化,简化数据挖掘流程。上述所有功能均基于 R 语言开发,并集成至网页服务器中,便于用户在线交互操作。我们为该平台整理了23个辅助数据库,涵盖暴露组、疾病及其相关的生物通路和分子,用于构建系统交互网络。网络节点包括:119,247,057个独特暴露物、17,186种疾病、153,780种蛋白质、19,122种代谢物、572,278条注释通路及43,324个基因本体(GO)术语。网络边包含十种交互类型,如暴露-蛋白质(3,971,005对)、暴露-GO(28,632对)、暴露-通路(572,278对)、蛋白质-疾病(10,153对)、基因-疾病(108,298,474对)、GO-疾病(3,085,406对)、通路-疾病(612,319对)以及用于荟萃分析和综述的暴露-疾病关联(1,244对)等。ExposomeX 能有效支持多维暴露组数据的分析,探索“暴露–生物学–疾病”之间的关联强度,增强对统计机制和生物机制的理解,提升预测能力,并实现基于高质量文献的荟萃分析自动化。通过对三个典型多组学数据集的重新分析验证,结果证明 ExposomeX 是一个高效工具,能够促进新型关联的发现并支持深入的生物网络研究。该新开发的分析框架支持全面的暴露组分析,用户可通过 R 程序或在线交互平台(http://www.exposomex.cn/)进行访问和使用
论文类型:长篇
第一作者:王斌(北京大学)
通讯作者:方明亮(复旦大学)
期刊:Environmental Science & Technology,IF2024 = 11.3,中科院1区
论文信息:Bin Wang, Changxin Lan, Guohuan Zhang, Mengyuan Ren, Tianxiang Wu, Ning Gao, Weinan Lin, Yanqiu Feng, Han Zhang, Bahabake Jiangtulu, Yuting Wang, Shu Su, Zhijian Liu, Xuqiang Shao, Fanrong Zhao, Bo Peng, Xiaotong Ji, Xiaojia Chen, Min Nian, Junjie Yang, and Mingliang Fang*. ExposomeX: Development of an Integrative Exposomic Platform to Expedite Discovery of the “Exposure−Biology−Disease” Nexus. Environmental Science & Technology, 2025, doi.org/10.1021/acs.est.5c05956
全文链接:https://pubs.acs.org/doi/10.1021/acs.est.5c05956

环境暴露在影响人类健康方面起着至关重要的作用。近年来,围绕混合暴露的风险评估策略已逐步建立,并融合了流行病学和实验研究证据。作为一种旨在揭示环境风险因素与多种疾病之间联系的新策略,暴露组学已成为下一代环境健康研究的核心焦点。在健康风险评估中,暴露组的概念涵盖了从受孕开始贯穿整个生命周期的所有环境暴露,强调对多源环境因素的全面考量。暴露组研究通常需要利用多维数据,涵盖从几个到成千上万个外部环境因素,这些因素可能来源于物理、化学和生物暴露,亦可能与社会行为相关。尽管暴露组在概念上极具吸引力,其发展仍受到诸多挑战的制约,例如研究设计复杂、分析能力受限以及大规模暴露测量的高成本等。因此,相较于已快速发展的基因组学、蛋白组学和代谢组学,暴露组学在特征化和系统分析方面仍处于起步阶段,急需建立标准化的分析流程。迄今为止,尚缺乏一个能够满足上述需求的整合型暴露组平台。然而,由于暴露组数据中存在高度多重共线性,现有统计方法难以高效地从相关暴露中识别出真正影响健康结果的关键因素,同时也难以处理暴露分类错误和识别潜在的协同作用。目前,暴露组研究的进一步发展面临三大主要挑战:
(1)整合和解释高维数据(包括外部暴露、内在生物反应以及疾病结局),并建立它们之间的关联。暴露组研究涵盖多种流行病学研究设计,如横断面、病例对照、队列、纵向和面板研究等,每种设计都对应不同的数据预处理与统计分析方法。此外,不同统计方法可能产生显著不同的结果,因此需要方法学上的交叉比较,以获得稳健可靠的结论。目前虽有如 rexposome 和 omicRexposome 等R语言工具包可供使用,但针对不同研究设计、数据结构和任务场景建立统一的统计分析流程仍属空白,特别对初学者而言尤为重要。环境全组关联研究(EWAS)已被证实在评估暴露组与健康的相关性方面具有良好的特异性和敏感性,但其主要关注于关联强度。进一步的深入分析,如中介效应、交互效应以及预测能力,在满足前提条件的情况下也应被纳入,以充分挖掘暴露组数据的潜力。
(2)构建外源性暴露、体内生物反应与疾病之间的生物机制内在联系。当前尚无平台能够系统整合并解释环境暴露、内源生物学变化与疾病之间的复杂关系。近年来,多个高质量数据库的建立为实现这一目标奠定了基础,例如药物、毒物和其他化学物质的生物靶标数据库,如比较毒理基因组数据库(Comparative Toxicogenomics Database,CTD)、蛋白与蛋白交互作用数据库(StringDB)、化学-蛋白互作网络数据库(STITCH)等。这些资源为构建一个能将暴露组结果与其他组学数据生物学地整合的平台提供了可行性。这些知识图谱的构建,对于排除混杂因素并提出可验证的高质量假设至关重要。
(3)亟需以健康结局为导向高效筛选与鉴定高分辨质谱检测得到的物质信号。在现有技术中,非靶向分析(NTA)因其高覆盖率与高通量的优势,被认为是暴露组研究中极具前景的方法之一,尽管其数据分析仍是主要瓶颈。与代谢组学或蛋白组学相比,暴露组分析所涉及的环境因素来源广泛,结构多样,仅靠传统的 t 检验、方差分析等方法进行特征筛选远远不够,因此,必须引入更复杂的统计模型来识别并优先考虑与目标疾病结局显著相关的质谱特征。此外,前文所述,不同统计方法可能得出不同的结果,因此结合多种分析方法并进行模型间的交叉验证尤显重要。为应对上述挑战,我们构建了一个整合型平台(ExposomeX),旨在标准化暴露组研究流程,并打通“暴露–生物学–疾病”的关键连接,为深入开展暴露组学研究提供系统化解决方案。
平台设计
ExposomeX旨在构建“暴露–生物学–疾病”(Exposure–Biology–Disease)之间的桥梁(见图1)。作为首个面向暴露组研究的综合分析平台,ExposomeX 包括网页版在线交互界面(www.exposomex.cn)(图1A)和对应的 R 语言分析包(https://github.com/ExposomeX)。该平台由六大核心功能模块构成:统计分析(E-STAT)、暴露组数据库(E-DB)、质谱数据处理(E-MS)、荟萃分析(E-META)、生物学关联(E-BIO)和数据可视化(E-VIZ)。这六个功能模块之间具有如下互联逻辑(图1B):E-DB 为 E-MS、E-BIO、E-META 和 E-STAT 提供基础信息支持,包括环境暴露、化学毒性、交互网络、统计模型及与多种疾病相关的效应量;E-MS 提供注释后的特征信息,用于 E-STAT 构建统计模型,并支持生物信息分析,识别非靶向分析(NTA)等暴露组关键特征用于生物解释;E-STAT 提供暴露组研究常用的统计工具,并可生成用于 E-MS 和 E-BIO 的输入内容,用于机制解析或新发现的验证;E-BIO 提供潜在的生物学关联,用于指导 E-STAT 和 E-MS 中统计模型的选择,助力构建“暴露–生物学–疾病”之间的因果路径;E-META 为 E-BIO 和 E-STAT 提供荟萃分析结果,增强统计解释能力;E-VIZ 基于 E-META、E-BIO、E-MS 和 E-STAT 的分析结果,提供探索性分析可视化功能。上述六大功能由15个已开发模块支撑,包括:E-STAT:StatTidy、StatDesc、StatCros、StatMO、StatPanel、StatSurv、StatMedt、StatMix、StatHEP、StatIP(共10个);E-DB:EDb;E-MS:EMS;E-META:EMeta;E-BIO:EBio;E-VIZ:EViz。平台使用的介绍视频已上传至 YouTube 和哔哩哔哩网站(图1C)。相关操作说明材料可通过官方网站下载(www.exposomex.cn/#/download)(图1D).

图 1. ExposomeX 平台设计。(A)基于网页的用户界面主页面;(B)平台六大功能之间的交互关系,箭头表示从起始功能到结束功能的支持作用,不同颜色区分各功能之间的连接;(C)包含 15 个支撑模块(隶属六大功能)的操作视频教程界面;(D)下载 23 个已整理支撑数据库的界面。
E-DB:数据库构建与应用
ExposomeX 平台六大功能模块的开发,首先依赖于系统构建的数据库资源,这些数据库涵盖了暴露化学物的检索、暴露生物标志物信息、化学物靶向蛋白、化学物–健康结局关系以及与疾病相关的蛋白质信息。这些数据库大多基于权威来源(如 CTD、STITCH、ToxCast 和 T3DB)进行汇总、清洗和交叉验证。此外,还手动检索了与非化学环境暴露因素相关的靶蛋白信息。关键信息整理于表1,数据库检索功能汇总见图2A。节点类信息(Node)数据库包括:
1) DB_Exposure:包含 119,247,057 个独立暴露条目,提供化学别名、IUPAC名称、CID、DTXSID、CAS号(CASRN)、InChIKey 和 SMILES(如可用);
2) DB_Biomarker:收录 2,050 个暴露生物标志物;
3) DB_Disease:包含 17,186 个疾病ID,以“EXD”前缀标识;
4) DB_Protein:收录 153,780 个蛋白质,以人类蛋白为主,少量涉及大鼠/小鼠等;
5) DB_Enzyme:来自 KEGG 的 5,221 个酶信息;
6) DB_Gene:覆盖所有研究物种,包含 51,607,653 个相关基因;
7) DB_Metabolite:来源于 KEGG 的 19,122 个人类代谢物;
8) DB_Pathway:主要来源于 CTD,包含 572,278 条与人类相关的通路;
9) DB_GO:包含 43,324 个GO术语。
关联类信息(Link)数据库包括:
1) DB_Exposure_Gene:包含 561,510 对暴露–基因关联
2) DB_Exposure_GO:包含 28,623 对暴露–GO关联
3) DB_Exposure_Pathway:包含 572,278 对暴露–通路关联(主要来源于 CTD)
4) DB_Exposure_Protein:包含 3,971,005 对暴露–蛋白互作信息,整合多个权威数据库
5) DB_Gene_Protein:来自 UniProtKB,包含 561,510 对基因–蛋白关联
6) DB_Protein_Protein:基于 STRING 数据库,收录 220,355 对人类蛋白–蛋白互作(联合评分 >500)
7) DB_Protein_Disease:包含 10,153 对蛋白–疾病关联
8) DB_Gene_Disease:包含 108,298,474 对人类基因–疾病关联
9) DB_GO_Disease:包含 3,085,406 对GO术语–疾病关联(来自 CTD)
10) DB_Pathway_Disease:包含 612,319 对通路–疾病关联(来自 CTD)
11) DB_Meta:收录 1,244 对用于荟萃分析或文献综述的暴露–疾病关联
这些数据库为 ExposomeX 各功能模块提供了强有力的数据支持,是实现暴露–生物–疾病关联建模与多维数据集成分析的核心资源。
表 1. ExposomeX平台数据库的主要特征及其数据来源
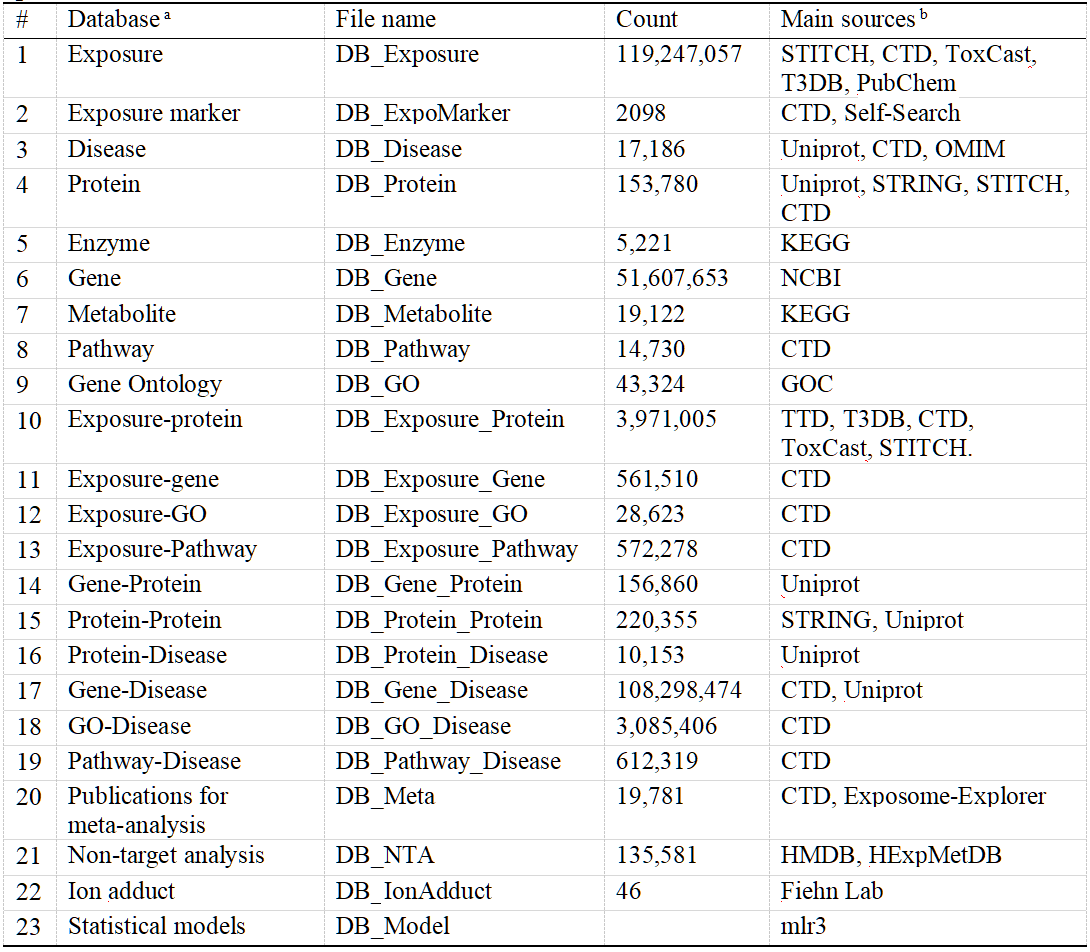
a数据库下载网址 (http://www.exposomex.cn/#/download);
E-META与E-BIO:对“暴露–生物学–疾病”关联的综合探索
在当前版本的 E-BIO 功能中,平台提出了多种典型的关联模式以整合“暴露–生物学–疾病”(Exposure–Biology–Diseases)之间的复杂关系(见图2B),包括 EGoD(暴露–GO–疾病)、EGeD(暴露–基因–疾病)、EPaD(暴露–通路–疾病)、EPD(暴露–蛋白–疾病)和 EPPD(暴露–蛋白–蛋白–疾病)等。例如,研究使用 EPPD 模型对13种环境暴露与6种疾病之间的生物网络关系进行了分析(图2C)。在该案例中,多个环境暴露物被假设作用于相同或相关的蛋白靶点,并与疾病产生关联,这表明可能存在共同作用机制或暴露–暴露之间的相互作用。在该假设网络中,杀虫剂敌敌畏(diazinon)与2,3,7,8-TCDD(二恶英)被识别为关键毒物,其潜在致病通路分别为:“Diazinon → EFNA2 → KIT → 睾丸生殖细胞肿瘤”以及“2,3,7,8-TCDD → PLAT → IL-6 → 类风湿性关节炎”,可用于机制假设或进一步验证。其中,EGoD 网络分析揭示了金属毒物诱导神经管缺陷(NTDs)的可能机制。所有涉及的金属毒物均已在既往流行病学研究中与NTDs相关联,并可较为明确地推测其作用通路与受损的蛋白/代谢途径。总体而言,该平台支持从暴露到疾病之间可能机制的可视化构建与网络映射,其代码与结果可在补充材料“图2的补充代码与数据”中查看。在 E-META 功能中:可通过MetaRef 子模块总结既往流行病学研究(见图2D);可使用MetaRev 探索特定暴露–疾病的知识总结,例如锌摄入与哮喘风险的关系(图2E);可通过MetaAsso 子模块对铜暴露与多囊卵巢综合征(PCOS)风险之间的关联进行荟萃分析(纳入12项研究)。该分析结果以森林图展示,并使用固定效应模型与随机效应模型进行汇总(图2F),并考虑各研究的权重贡献。平台还提供交互式功能,允许用户上传新的研究文献,持续扩展荟萃分析数据库。用户可通过社区页面(http://www.exposomex.cn/#/community)下载标准化表格并上传信息,以便系统集成与更新分析结果。
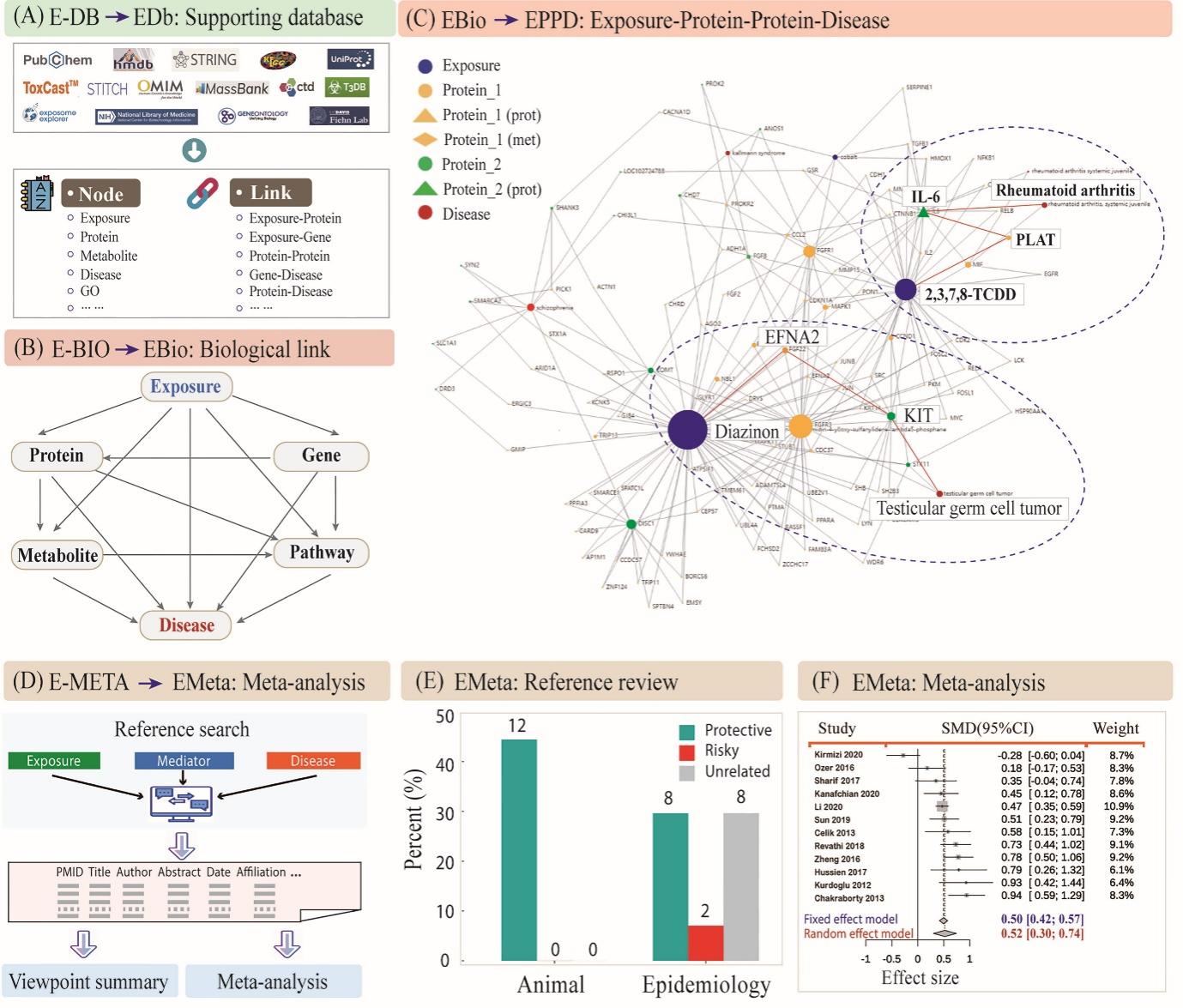
图 2. 构建环境暴露、生物通路与健康结局之间的生物学关联。(A)E-BIO 功能中综合数据库及其检索框架;(B)利用 E-BIO 模块构建环境暴露与多种疾病之间生物学关联的框架;(C)E-BIO 中“暴露-蛋白-蛋白-疾病”(EPPD)模式的应用示例,圆圈大小代表该节点的连边数,虚线圈标示典型的暴露-疾病关联;(D)基于数据挖掘技术和既有文献数据库(E-META)构建环境暴露与健康结局直接关联的框架;(E)E-META 中关于锌摄入与哮喘风险关联文献回顾的应用示例;(F)基于荟萃分析前人关于铜暴露与多囊卵巢综合征风险关联研究的示例。
E-MS:从NTA数据中筛选与健康结局显著相关的特征
E-MS 功能可高效识别并标注来自不同流行病学研究设计中的正向或负向关联特征(见图3A)。例如,在一个以变量“Y1”为健康结局的队列研究中,E-MS 共分析了 6,552 个特征(图3B)。在进行多重检验校正后,仅有 58 个特征 被识别为与健康结局显著相关,从而有效地在庞大的特征数据中完成优先级排序与注释筛选。对于非靶向分析(NTA)数据,本平台旨在构建结果导向的统计方法,借助 E-MS 提供的 MS/MS碎片信息(即 MS2),识别已注释的重要特征(图3C),并支持后续的深入验证分析。
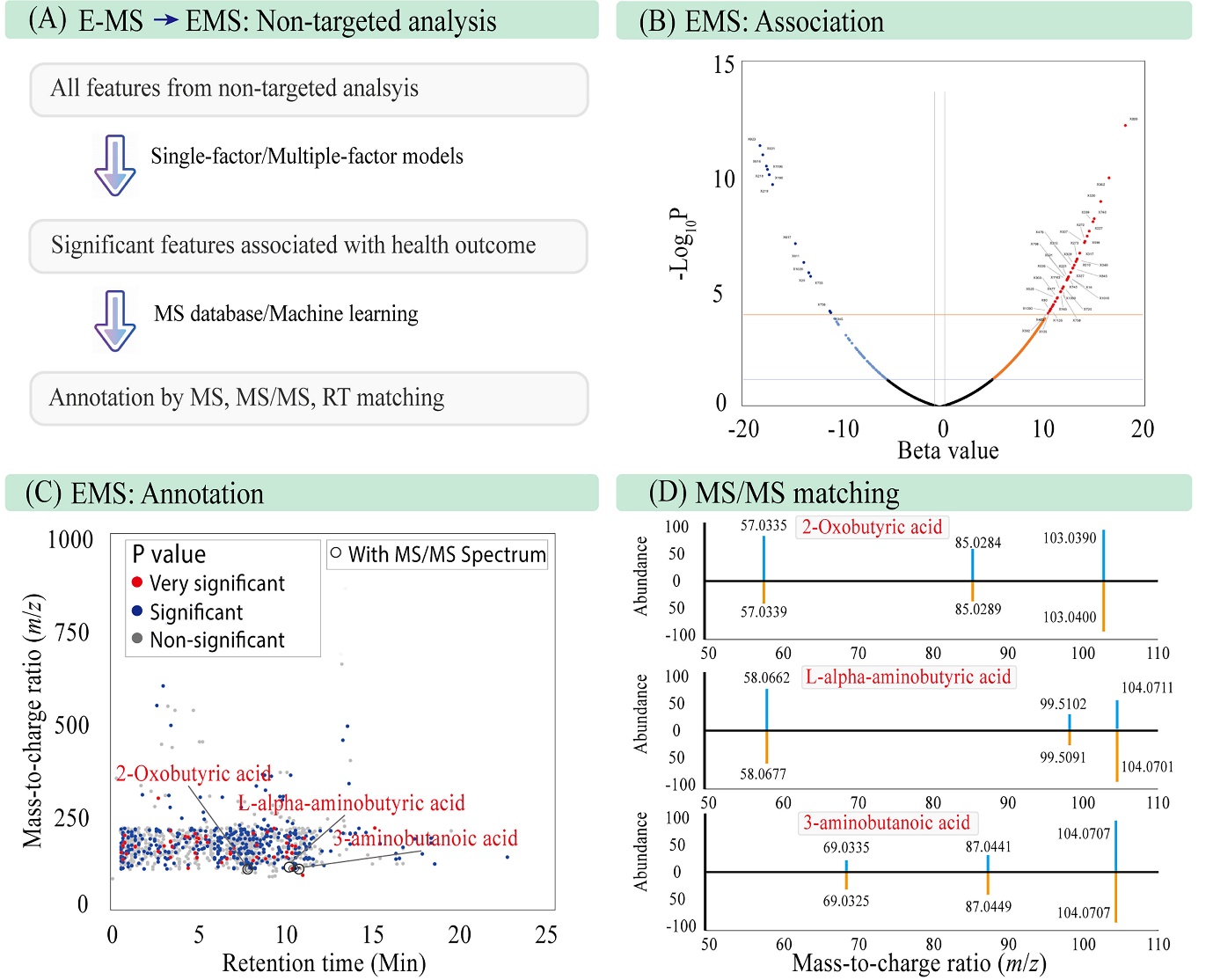
图 3. 基于 MO-Study 多组学数据构建 E-MS 非靶向分析功能。(A)网页界面设计;(B)显著特征的火山图;(C)利用 MS/MS 信息注释的特征;(D)三种化合物的 MS/MS 匹配结果。
E-STAT:暴露组分析框架与流程的标准化
我们进一步开发了暴露组研究的数据分析框架与标准化操作流程(见图4A)。在建模任务中,E-STAT 提供了多个功能模块,分别适用于以下四类分析任务:
1) 关联分析:如 StatCros、StatPanel、StatSurv、StatMedt、StatMix
2) 预测分析:如 StatCros、StatHEP
3) 多组学整合分析:StatMO
4) 统计解释与贡献度评估:StatIP
具体选择哪个模块,需结合研究的流行病学设计、数据结构及分析任务类型综合判断。目前,ExposomeX 支持多种常见的研究设计,包括横断面研究、纵向研究、病例对照研究、队列研究以及病例随访等,并匹配相应的数据结构(如横断面数据、面板数据、生存数据)。平台进一步引入多种主流机器学习算法,在传统建模方法(如线性回归、正则化回归、贝叶斯方法、核函数方法、样条函数算法)基础上,拓展了集成模型(如决策树、人工神经网络、Boosting、Bagging 和堆叠集成 Stacked Generalization, SG),以实现更优预测效果。
在图4B–G中展示了多个典型模型的性能。以下为主要分析结果:效应量评估:通过 StatCros、StatPanel、StatSurv、StatMedt 和 StatMix 模块评估多种研究设计中暴露–结局的正负相关性。例如,在某队列研究中,StatCros 分析了 6,552 个特征 与健康结局的关联(图4B)。混合暴露分析:StatMix 提供多种主流方法,包括多因素逐步线性回归(MLR)、LASSO、弹性网(ENET)、加权分位和回归(WQS)、分位 G-计算(G-Comp)与贝叶斯核机器回归(BKMR),用户可据此评估混合暴露效应及其交互作用,部分模型揭示了明显的剂量-反应效应(图4C)。中介效应分析:本平台引入多变量中介机制框架,结合环境风险评分构建与中介路径探索(图4D),支持配对中介分析、暴露维度降维、中介变量压缩与惩罚项回归。预测分析:StatCros 用于单组学预测,StatMO 用于多组学预测(图4E, 4F)。其中,StatCros 支持不同模型的 ROC 曲线可视化;StatMO 示例中采用 SG 模型整合多个子模型的预测结果(暴露组、代谢组、蛋白组、免疫组)。降维与网络构建:平台提供两种降维策略:变量正则化选择与变量重要性筛选。可构建组内(intra-omics)及组间(inter-omics)网络,用于识别潜在的生物学关联,提升结果的可靠性与可重复性。统计解释与贡献度评估:通过 StatIP 模块评估暴露变量的贡献度及其剂量-反应趋势,例如图4G中选取贡献度最高的变量 X21 进行展示。
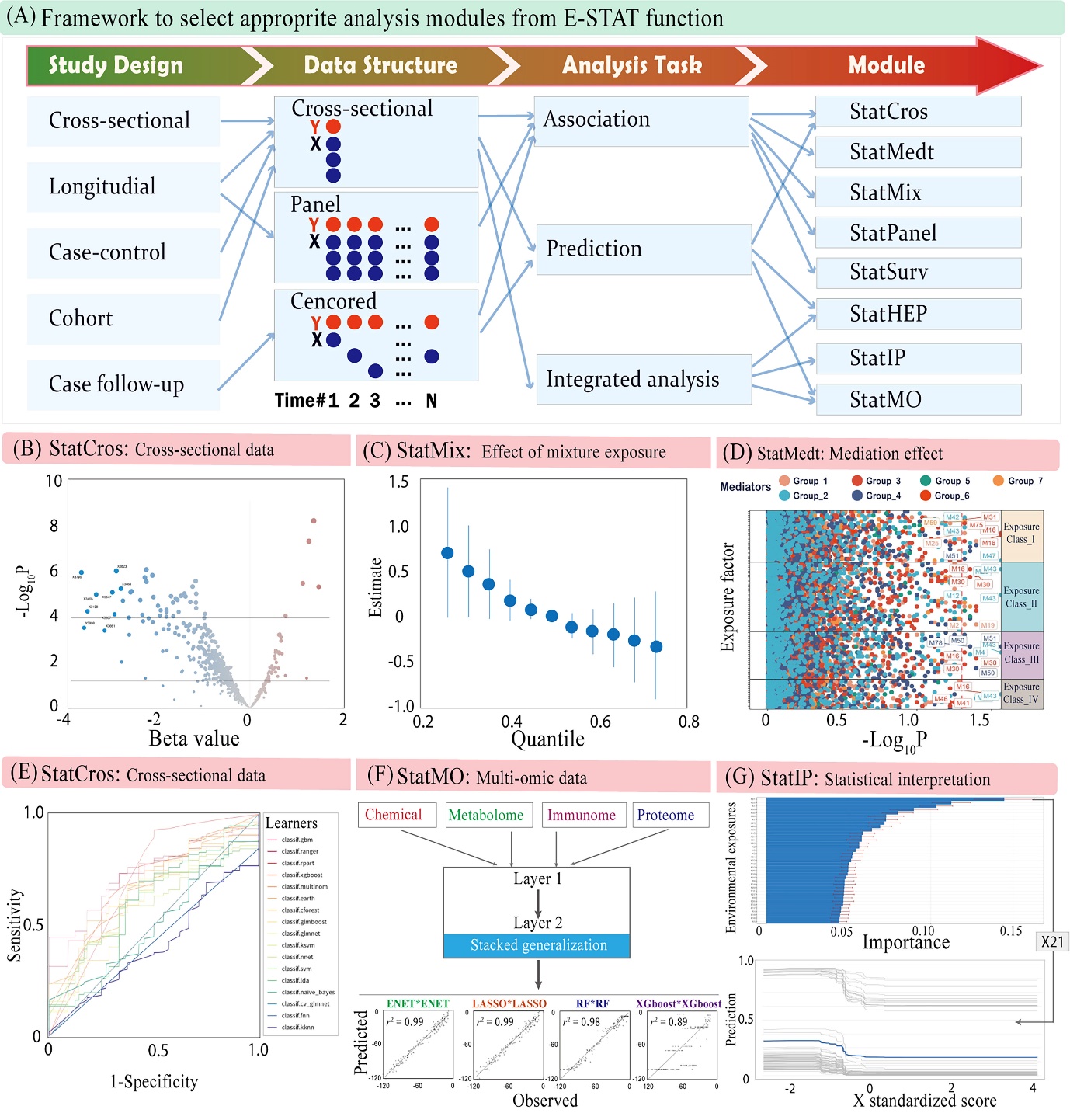
图 4. 为暴露组数据构建统计模型。(A)在流行病学研究设计、数据结构与分析任务的综合考量下选择合适模型的框架;(B)使用 StatCros 进行关联分析的效应量,“Beta 值”表示线性回归模型系数;(C)使用 StatMix 探索多种环境暴露对健康结局的混合效应;(D)使用 StatMedt 在四类潜在暴露组与七类潜在中介组间筛选中介变量;(E-F)分别使用 StatCros 与 StatMO 对单组学与多组学数据进行预测分析;(G)使用 StatIP 对典型因素(如 X21)的偏依赖曲线分析,变量重要性和剂量–反应趋势基于 R 包 “DALEX” 及随机森林算法计算。
ExposomeX 的独特优势与对比分析
自2005年“暴露组”概念提出以来,其受到了广泛关注,这是因为人们普遍认识到环境暴露在全球疾病负担中占据重要地位。因此,暴露组的核心目标是为解析环境暴露对健康的真实影响提供新的视角。然而,截至目前,暴露组学的成功实践案例仍然较为有限,相关文献多为观点性文章或综述。与代谢组学、蛋白组学等其他组学领域相比,暴露组研究在研究设计和数据分析上面临更大的挑战。其主要困难包括:如何整合并解释高维暴露数据;理解外源性暴露与疾病之间的生物学机制;从非靶向分析(NTA)中识别与疾病相关的显著特征;有效整合多源数据库和可视化工具等。此外,暴露组学本身具有高度多学科交叉性,需要流行病学家、临床医生、统计学家、化学家、地理学家、经济学家、社会学家与毒理学家的共同参与。由于缺乏系统的统计背景支持,研究中存在误用模型的风险,进而可能影响结果的准确性。与此同时,不同统计方法可能给出不同的分析结果,若仅依赖单一方法,容易遗漏暴露组数据中潜在的重要信息。为应对这些挑战,我们开发了ExposomeX平台,旨在打破暴露组研究中存在的技术壁垒与分析瓶颈,推动该领域向标准化、系统化方向发展。
ExposomeX 的目标并非“重复造轮子”,而是在已有研究基础上提出了多个创新点,不仅涵盖了数据库的系统整合与清洗、分析方法的开发,还包括多种数据库与算法的融合,最终构建了一个可加速暴露–生物–疾病关系发现与分析的高效平台。ExposomeX 与其他已有平台(如 Exposome-Explorer、rexposome 项目、Exposome-NL、HMDB、T3DB 和 Human Blood Exposome DB)之间的异同点,具体见表2。总体而言,ExposomeX具备支撑数据库、NTA分析、统计模型选择、生物学解释、荟萃分析、数据可视化、用户交互(R包和网页操作、数据互动等)。
表2. ExposomeX与其他暴露组相关平台特征对比情况
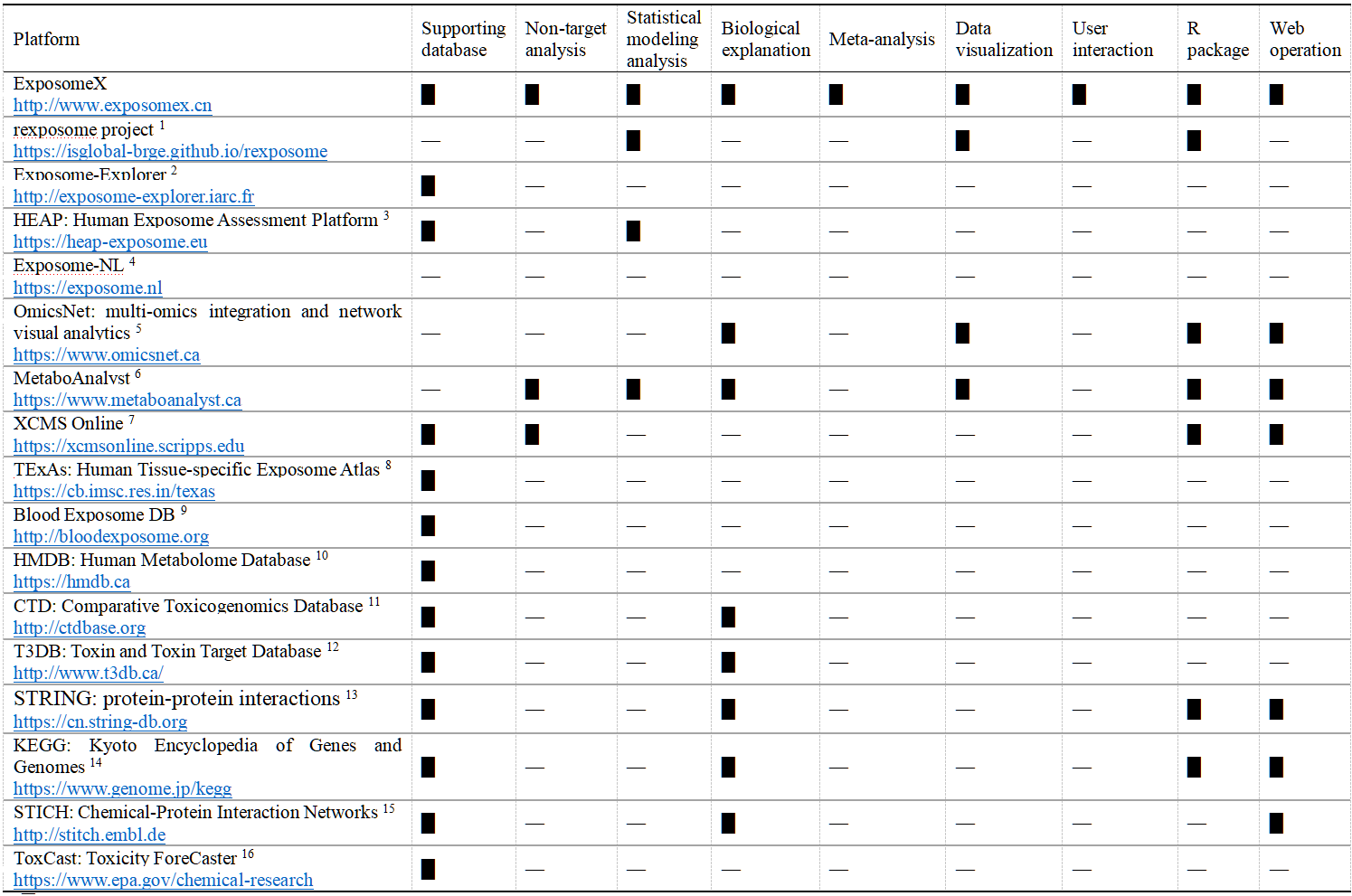
“█” 提供信息; “—”未提供信息
从科学性与应用性两方面来看,ExposomeX 都具有开创性意义:
1) 科学方面:ExposomeX 构建了目前整合度最高的暴露组学分析框架,覆盖统计分析与生物学解释两大方向,支持用户深入挖掘暴露组数据中隐藏的重要信息,尝试性地建立“暴露–生物–疾病”网络关系。相比于以往多数仅关注多组学相关性的研究,ExposomeX 首次系统构建并分析该生物网络。平台支持多种统计建模方式,能够在同一重采样数据集上对比多种模型的性能,开展多任务分析,确保发现的新信息更加客观、可重复、无偏倚。ExposomeX 汇集、清洗并整合了最大规模的暴露组研究数据库,涵盖 CTD、ToxCast、STITCH 等权威资源,并补充了包括 PM2.5、睡眠障碍、辐射等关键环境暴露的信息,极大增强了平台的数据深度与广度,如 E-META 和 E-MS 等创新模块进一步提升了平台的分析能力。
2) 在应用层面,ExposomeX 是首个同时提供高交互性用户界面的暴露组平台,支持开放的 R 包与网页版平台双通道访问;用户可完全获取 R 源码与数据库,并可根据需要修改数据库内容——这是其他平台(如 STITCH)普遍不具备的功能;网页端平台支持高效的云端分析与结果共享,用户还可上传研究结果,丰富数据库内容,促进暴露组研究社区的发展。此外,平台各模块间高度联通,模块输出可直接作为其他模块的输入,使分析流程高度简化,有助于通过代码实现全流程自动化与可重复性分析。
通过对三项代表性研究——中介分析(Medt-Study)、多组学整合分析(MO-Study)和早产预测(SPB-Study)的重新分析,平台展现了其在暴露组整合分析中的巨大潜力。特别是在 SPB-Study 重分析 中,仅使用了 60 名女性的数据,就成功演示了如何结合机器学习方法,从多个环境暴露物中筛选关键分子事件,有效体现了 ExposomeX 在数据挖掘方面的显著优势。目前该平台已经过去中国国家版权局颁发的计算机软件著作权登记证书(图5)。

图5. ExposomeX软件著作权证书
总结与展望:
ExposomeX 是一个集成了 R 包与网页版交互功能的生态型平台,其统一的设计理念、语法规范与数据结构,为暴露组数据的处理与分析提供了一个全面、透明、可追溯、可复现、面向对象的计算框架。ExposomeX 的建立将为暴露组研究领域带来显著推动作用,尤其体现在以下几个方面:标准化分析框架的构建、数据共享的便利化、分析结果的可重复性保障。平台设计核心强调的灵活性与可扩展性,使其能够无缝集成其他研究工具,从而提升其在暴露组学研究社区中的适应性与延展性。未来版本将不断更新统计方法,并拓展数据库种类,进一步增强平台在暴露组学研究中的适用能力。同时,平台还将鼓励用户参与扩充荟萃分析的文献数据库,推动社区协作。这些举措将共同促进构建一个稳健的平台,使暴露组研究能够真正实现标准化、实用化与可操作化。
致谢:
感谢中国队列共享平台环境暴露与人类健康工作组(http://chinacohort.bjmu.edu.cn)提供合作平台。特别感谢来自华北电力大学刘志坚教授、邵绪强教授及其团队研究生(李明宇、李百强、金艺中和张思琦)在平台网页界面开发中所付出的技术支撑!同时,也感谢复旦大学的赵峰、王思懿、杨婧,以及来自新加坡南洋理工大学的刘敏博士、赵昊铎和杜慧丽在数据库构建与方法开发中的积极参与。此外,感谢北京大学的同学参与平台代码和网页测试工作。项目所有参与者请见网站:http://www.exposomex.cn/#/about
本研究得到了以下项目的资助支持:中国科技部国家重点研发计划(项目编号:2023YFC3708305,2022YFE0134900,2022YFC3702600,2024YFA0918901);中国科学院战略性先导科技专项B类——新污染物项目(编号:XDB0750300);国家自然科学基金项目(编号:42077390,41771527)。

